Capítulo 6 Desequilíbrio de Ligação
.](https://acsess.onlinelibrary.wiley.com/cms/asset/72da5c7a-6c96-450d-b9d1-20d9e0bad237/tpg2plantgenome2018060044-fig-0003-m.jpg)
Figura 6.1: O desequilíbrio de ligação decai com a distância física entre as regiões gênicas, como mostrado pela correlação (r2) nos diversos cromossomos do sorgo (Sorghum bicolor). Perceba que raças tradicionais (RT, Landraces) apresentam uma taxa de decaimento maior do que linhagens endocruzantes recombinantes (LER, RILS). Ver Hu et al 2019.
Linhagem Endocruzante Recombinante – Um organismo com cromossomos que incorporam um conjunto essencialmente permanente de eventos de recombinação entre cromossomos herdados de duas ou mais linhagens endocruzantes.
Raça Tradicional – Uma variedade tradicional domesticada e adaptada localmente de uma espécie de animal ou planta que se desenvolveu ao longo do tempo, por meio da adaptação ao seu ambiente natural e cultural de agricultura ou pastorícia, e devido ao isolamento de outras populações da espécie.
6.1 Ligação gênica
A ligação gênica é a tendência de genes localizados no mesmo cromossomo serem herdados em conjunto durante a meiose.
Genes ditos estarem geneticamente ligados correspondem a loci que estão perto um do outro, sendo menos susceptíveis de ser separados em diferentes cromátides por recombinação genética.
Considera-se recombinação genética toda e qualquer produção de uma nova combinação de alelos em dois ou mais loci gênicos. Em organismos diplóides, isto se dá principalmente por meio do mecanismo de crossing over.
Assim sendo, a proporção de descendentes que sofreram um evento de recombinação entre dois loci gênicos é medida pela taxa de recombinação genética (\(r\)).
Desta forma que, os descendentes herdarão cromossomos:
iguais ao de um dos parentais, em uma taxa \(1 − r\); ou
que são uma recombinação dos cromossomos dos parentais, em uma taxa \(r\).
Uma vez que a recombinação por crossing over só ocorre entre os dois cromossomos homólogos pareados da tétrade na meiose, o maior valor que a taxa de recombinação genética pode alcançar é igual a \(1/2\), que é igual o mesmo que chance de encontro de alelos em loci em cromossomos diferentes, i. e., segregando independentemente.
Figura 6.2: Medição empírica da taxa de recombinação – Linhagens parentais (\(F_0\)) puras são cruzadas para produzir indivíduos heterozigotos no dois loci gênicos (diíbridos), cuja variação entre os alelos esteja ligada nos cromossomos parentais (cis), os quais são cruzados com uma outra linhagem pura (testador) para quantificar os tipos parentais e recombinantes produzidos.
6.2 Frequências haplotípicas
Haplótipo – 1. coleção de alelos específicos em um agrupamento de genes fortemente ligados em um cromossomo, que provavelmente serão herdados juntos; 2. um conjunto de alelos de polimorfismo de nucleotídeo único (SNP) ligados que tendem a sempre ocorrer juntos, i.e., que estão associados estatisticamente.
Considere os haplótipos produzidos por dois loci gênicos \(A\) e \(B\), com dois alelos cada, com taxa de recombinação \(r\) entre si.
A gametogênese dos indivíduos do genótipo \(AB/ab\) produzirá os seguintes haplótipos:
| Haplótipo | Frequência | Tipo |
|---|---|---|
| \(AB\) | \((1-r)/2\) | Parental |
| \(ab\) | \((1-r)/2\) | Parental |
| \(Ab\) | \(r/2\) | Recombinante |
| \(aB\) | \(r/2\) | Recombinante |
Enquanto que a gametogênese de um genótipo \(Ab/aB\) produzirá os seguintes haplótipos:
| Haplótipo | Frequência | Tipo |
|---|---|---|
| \(AB\) | \(r/2\) | Recombinante |
| \(ab\) | \(r/2\) | Recombinante |
| \(Ab\) | \((1-r)/2\) | Parental |
| \(aB\) | \((1-r)/2\) | Parental |
Sendo a frequência observada dos haplótipos dos loci gênicos \(A\) e \(B\):
| Haplótipo | Frequência |
|---|---|
| \(AB\) | \(p_{AB}\) |
| \(ab\) | \(p_{ab}\) |
| \(Ab\) | \(p_{Ab}\) |
| \(aB\) | \(p_{aB}\) |
De forma que,
\[p_{AB} + p_{ab} + p_{Ab} + p_{aB} = 1\]
Note que estas são frequências relativas, que determinam as frequências alélicas:
| Alelo | Frequência |
|---|---|
| \(A\) | \(p_{A} = p_{AB} + p_{Ab}\) |
| \(a\) | \(p_{A} = p_{ab} + p_{aB}\) |
| \(B\) | \(p_{A} = p_{AB} + p_{aB}\) |
| \(b\) | \(p_{A} = p_{ab} + p_{Ab}\) |
Acoplamento gênico – Quando ambos os alelos do tipo selvagem estão em um cromossomo homólogo, e ambos os alelos mutantes estão no outro (e. g., \(AB/ab\)). Também conhecido como configuração cis.
Repulsão gênica – Quando um alelo do tipo selvagem e um alelo mutante estão em um cromossomo homólogo, e o oposto no outro (e. g., \(Ab/aB\)). Também conhecido como configuração trans.
6.3 coeficiente de desequilíbrio de ligação
O termo desequilíbrio de ligação (LD, linkage disequilibrium) foi usado pela primeira vez por Lewontin e Kojima (1960) para indicar uma associação não-aleatória de alelos em dois ou mais loci.
Dado suficiente tempo evolutivo…
a ocorrência de eventos de recombinação aleatória em um cromossomo resultará em uma distribuição de equilíbrio de alelos em cada locus; e
a frequência de um alelo em particular em um determinado locus será independente da frequência de alelos em outros loci ligados.
Nesse sentido, seria alcançado um equilíbrio de ligação, e a frequência do haplótipo entre um par de alelos será igual ao produto das frequências alélicas individuais, de forma que:
\[p_{AB} = p_{A} \cdot p_{B}\]
Assim sendo, o desequilíbrio de ligação é definido como a diferença entre a frequência observada de uma combinação particular de alelos em dois loci gênicos e a frequência esperada para a associação aleatória (esperada no equilíbrio).
\[D_{AB} = p_{AB} - p_{A}p_{B}\]
A medida \(D_{AB}\) é o coeficiente de desequilíbrio de ligação.
O coeficiente de desequilíbrio de ligação é definido para um par específico de alelos, no caso \(A\) e \(B\), e não depende de quantos outros alelos estão nos dois loci – cada par de alelos tem seu próprio coeficiente.
Na ausência de forças evolutivas, o coeficiente de desequilíbrio de ligação (\(D\)) convergirá para zero ao longo do tempo (atingindo o equilíbrio de ligação), e o tempo (\(t\)), em termos de número de gerações, que levará para tal convergência ocorrer irá depender da magnitude da taxa de recombinação (\(r\)) entre os dois loci.
Assim, a taxa de recombinação irá mudar a frequência do haplótipo da próxima geração (\(p_{AB}'\)) de forma que:
\[p_{AB}' = \left( 1-r \right) p_{AB} + rp_{A}p_{B}\]
onde \((1-r)\) e \(r\) são as chances de formação de haplótipos não-recombinantes e recombinantes, respectivamente.
A equação acima pode ser reescrita (viz., subtraindo \(p_{A}p_{B}\) dos dois lados) como:
\[p_{AB}' - p_{A}p_{B} = \left( 1-r \right) \left( p_{AB} - p_{A}p_{B} \right)\]
Sendo \(D_{AB}\) o coeficiente de desequilíbrio da geração atual, e \(D_{AB}'\) o coeficiente de desequilíbrio da próxima geração, temos então que:
\[D_{AB}' = \left( 1-r \right) D_{AB}\]
Desta forma, para qualquer par de alelos em dois loci gênicos ligados, de uma geração para a seguinte, temos:
\[D_{t+1} = \left( 1-r \right) D_{t}\]
E assim, partir desta, temos:
\[D_{t} = \left( 1-r \right)^t D_{0}\]
Logo, se \(t → ∞\), então \(D_t → 0\).
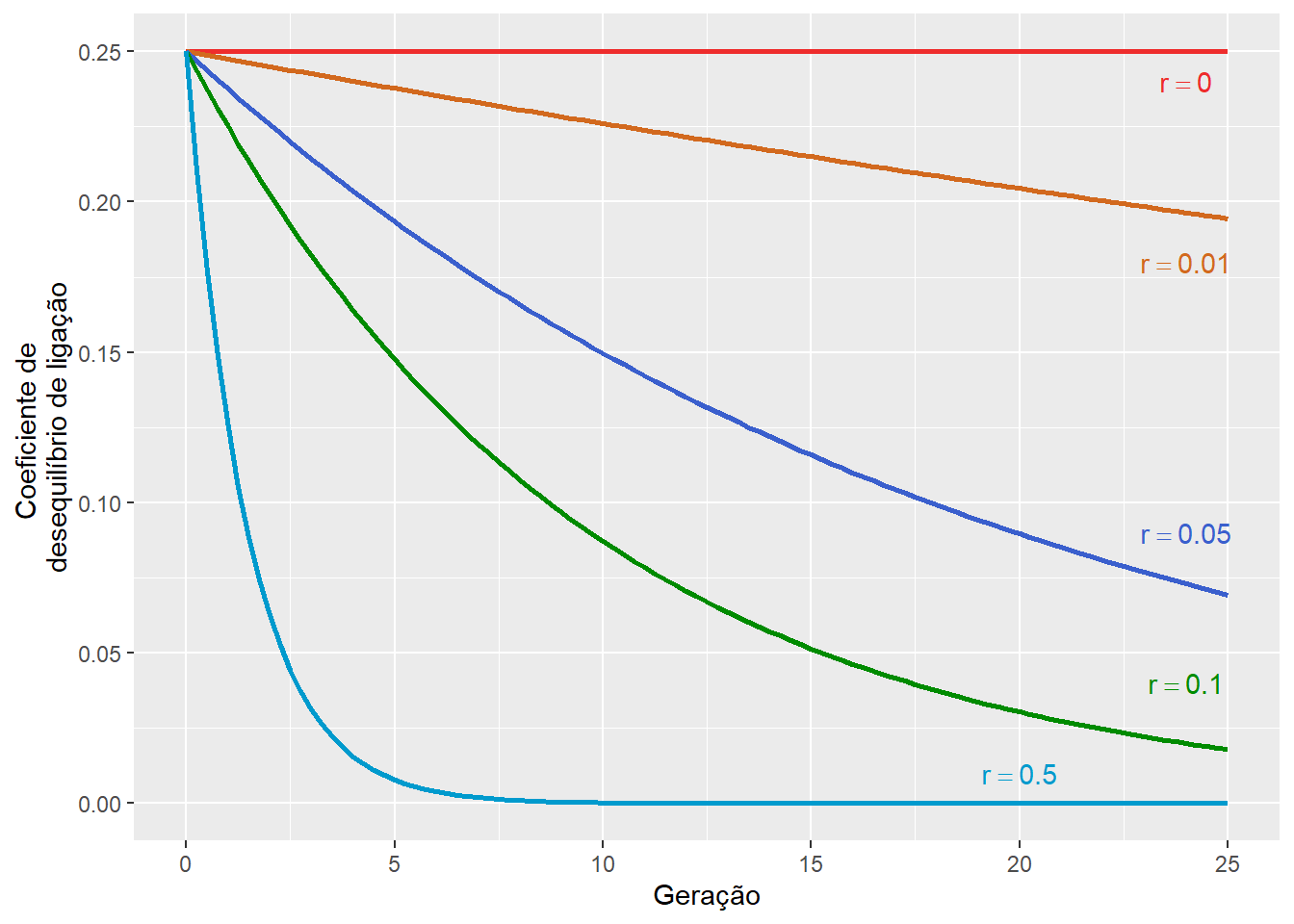
Figura 6.3: O coeficiente de desequilíbrio de ligação (\(D\)) ao longo das gerações sob diferentes taxas de recombinação (\(r\)).
O coeficiente de desequilíbrio de ligação é usado geralmente para indicar que dois genes estão fisicamente ligados.
No entanto, a detecção de um desequilíbrio de ligação não assegura a existência de ligação gênica ou falta de equilíbrio, podendo alternativamente refletir:
A ocorrência de LD pode refletir…
no genoma, como um todo:
histórias populacionais,
sistemas de acasalamento,
padrões de subdivisão geográfica
-e em regiões gênicas específicas:
- seleção natural,
- convergência gênica,
- mutação,
- e outras forças que promovem mudança nas nas freqüências gênicas.Como esses fatores afetam o equilíbrio de ligação entre um par particular de locos ou em uma região genômica depende das taxas locais de recombinação.
6.4 coeficiente de desequilíbrio de ligação normalizado
O coeficiente de desequilíbrio de ligação (\(D\)) nem sempre é uma medida conveniente, porque o seu intervalo de valores possíveis depende das frequências dos alelos a que se refere.
Isso dificulta a comparação do nível de desequilíbrio de ligação entre diferentes pares de alelos.
O coeficiente de desequilíbrio de ligação pode ser normalizado (\(D'\)) se dividindo-o pela diferença máxima teórica entre as frequências de haplótipos observadas e esperadas como:
\[\begin{equation} D’ = \frac{D}{D_{max}} \tag{6.1} \end{equation}\]
Onde,
\[\begin{equation} D_{max} = \min \begin{cases} \left \{p_A(1-p_B) , (1 – p_A)p_B \right \} & \text{se } D > 0 \\ \left \{p_Ap_B , (1 – p_A)(1 – p_B) \right \} & \text{se } D < 0 \\ \end{cases} \tag{6.2} \end{equation}\]
6.5 Coeficiente de correlação
Uma forma alternativa de expressar o desequilíbrio de ligação entre pares de alelos é por meio do coeficiente de correlação (\(r^2\)), expresso como:
\[r^2 = \frac{(D_{AB}^2)}{p_A(1-p_A)p_B(1-p_B)}\]
As medidas do coeficiente de correlação \(r^2\) e do coeficiente de correlação normalizado \(D'\) têm seus intervalos de valores limitados, e não abrangem todos os valores de \(0\) a \(1\) para todos os pares de loci.
O valor máximo de \(r^2\) depende das frequências alélicas nos dois loci sendo comparados, e só pode variar totalmente de \(0\) a \(1\) quando as frequências alélicas em ambos os loci são iguais, \(p_A = p_B\), se \(D_{AB} > 0\), ou quando as frequências alélicas têm a relação \(p_{A}=1-P_{B}\), se \(D_{AB} < 0\).
Por sua vez, \(D'\) sempre poderá ter um valor máximo teórico de \(1\), mas seu valor mínimo para dois loci será igual a \(| r |\) para esses loci.
Lewontin 1964. doi: 10.1093/genetics/49.1.49
Hill & Robertson 1968. 10.1007/BF01245622
Slatkin 2008. 10.1038/nrg2361
Smith 2020. doi: 10.1016/j.tpb.2020.02.005
6.6 Exercícios
6.6.1 Exercício 1 – Ligação gênica em Drosophila
O estudo de uma variação em um sítio de restrição \(EcoRI\) na região do gene álcool desidrogenase (\(Adh\)) em moscas-da-fruta capturadas em um mercado na Holanda (Cross & Birley 1986) revelou os seguintes dados:
| \(EcoRI^+\) | \(EcoRI^-\) | |
|---|---|---|
| \(Adh^F\) | 22 | 3 |
| \(Adh^S\) | 4 | 5 |
onde \(Adh^F\) e \(Adh^S\) são alelos da alozima \(Adh\) e \(Eco\)RI\(^+\) e \(Eco\)RI\(^-\) indicam, respectivamente, a presença ou ausência de um sítio de restrição \(Eco\)RI 3.5 kb à jusante do gene \(Adh\).
As duas regiões gênicas estão em equilíbrio de ligação?
Qual o coeficiente de desequilíbrio de ligação normalizado (\(D'\)).
6.6.2 Exercício 2 – Ligação gênica em SNPs
Considere os sequintes Polimorfismos de nucleotídeo único (SNPs) abaixo:
| SNP1 | SNP2 | SNP3 | SNP4 | |
|---|---|---|---|---|
| Indivíduo 1 | A/A | C/C | A/A | T/T |
| Indivíduo 2 | A/G | C/T | A/A | G/G |
| Indivíduo 3 | A/G | C/T | A/C | T/G |
| Indivíduo 4 | A/A | C/C | A/A | G/G |
| Indivíduo 5 | G/G | T/T | C/C | G/T |
Qual é o coeficiente de desequilíbrio de ligação normalizado (\(D'\)) entre esses loci gênicos?
Qual é o coeficiente de correlação (\(r^2\)) entre esses loci gênicos?