Capítulo 14 Seleção Natural
.](figs/linssetal2014.jpg)
Figura 14.1: O controle químico do mosquito Aedes aegypti vem dependendo principalmente de inseticidas piretróides, que interagem com o canal de sódio controlado por voltagem no sistema nervoso do animal. Mutações de substituição em sítios específicos da proteína conferem ao mosquito resistência ao inseticida, sendo estas conhecidas como mutações kdr (do inglês, knockdown resistance). Retirado de Linss et al (2014.
Soderlund & Knipple 2003. doi: 10.1016/S0965-1748(03)00023-7
Melo-Costa et al. 2020. doi: 10.1038/s41598-020-70029-7
14.1 Aptidão reprodutiva absoluta
Algumas diferenças podem melhorar as chances de um indivíduo sobreviver, se reproduzir e deixar mais descendes. Se os traços que dão a estes indivíduos uma vantagem adaptativa também são hereditários, estes serão passados dos genitores para os descendentes, o que levará a uma aptidão reprodutiva (ou fitness) diferencial. Mesmo que a vantagem adaptativa seja muito pequena, ao longo de muitas gerações, qualquer característica hereditária vantajosa tornar-se-á dominante na população.
Figura 14.2: Genótipos que conferem maior sucesso reprodutivo aos indivíduos tenderão a suplantar os de menor sucesso reprodutivo ao longo do tempo pelo mecanismo da seleção natural.
Ao modelar a seleção natural, a aptidão (ou fitness) reprodutiva de um genótipo se refere à sua contribuição para (ou a probabilidade de) sobrevivência (\(l\)), fecundidade e fecundabilidade (\(m\)), fertilidade (\(n\)) e viabilidade (\(o\)) dos indivíduos daquele genótipo.
Figura 14.3: .
Se considerarmos o potencial efeito desses parâmetros no sucesso repodutivo de cada genótipo formado pelos alelos \(1\) e \(2\) em uma dada geração \(t\), teremos:
| Genótipo | Número deindivíduos | Sobrevivência | Fecundidade | Fertilidade | Viabilidade |
|---|---|---|---|---|---|
| \(D(11)\) | \(p^2N_t\) | \(\color{blue}{l_{11}}\) | \(\color{green}{m_{11}}\) | \(\color{goldenrod}{n_{11}}\) | \(\color{red}{o_{11}}\) |
| \(H(12)\) | \(2pqN_t\) | \(\color{blue}{l_{12}}\) | \(\color{green}{m_{12}}\) | \(\color{goldenrod}{n_{12}}\) | \(\color{red}{o_{12}}\) |
| \(R(22)\) | \(q^2N_t\) | \(\color{blue}{l_{22}}\) | \(\color{green}{m_{22}}\) | \(\color{goldenrod}{n_{22}}\) | \(\color{red}{o_{22}}\) |
Assim, o número esperado de indivíduos de um determinado genótipo na próxima geração, dado o seu sucesso reprodutivo será…
… para o genótipo homozigoto dominante \(D(11)\):
\[ D_{t+1} = p^2 N_t \color{blue}{l_{11}} \color{green}{m_{11}} \color{goldenrod}{n_{11}} \color{red}{o_{11}}\]
… para o genótipo heterozigoto \(H(12)\):
\[ H_{t+1} = 2pq N_t \color{blue}{l_{12}} \color{green}{m_{12}} \color{goldenrod}{n_{12}} \color{red}{o_{12}}\]
… e para o genótipo homozigoto recessivo \(R(22)\):
\[ R_{t+1} = q^2 N_t \color{blue}{l_{22}} \color{green}{m_{22}} \color{goldenrod}{n_{22}} \color{red}{o_{22}}\]
E desta forma, a resultante do produto dos parâmetros \(l\), \(m\), \(n\) e \(o\) corresponderá ao valor da aptidão reprodutiva absoluta (\(W\)) da cada genótipo, onde:
\[\color{blue}{l_{11} m_{11} n_{11} o_{11}} = \color{blue}{W_{11}}\]
\[ \color{green}{l_{12} m_{12} n_{12} o_{12}} = \color{green}{W_{12}}\]
\[ \color{red}{l_{22} m_{22} n_{22} o_{22}} = \color{red}{W_{22}}\]
14.2 Aptidão reprodutiva relativa
As medidas de aptidão reprodutiva absoluta podem variar consideravelmente entre as diferentes linhagens evolutivas, sendo assim mais prático utilizar uma medida padronizada desses valores.
Ao dividir a aptidão reprodutiva absoluta de cada genótipo pela aptidão máxima observada entre todos os genótipos, convertemo-os assim em uma medida de aptidão reprodutiva relativa (\(w\)), definidos como:
\[\color{blue}{w_{11}}= \frac{\color{blue}{l_{11} m_{11} n_{11} o_{11}}}{\max\left(l_{ij} m_{ij} n_{ij} o_{ij} \right)} = \frac{ \color{blue}{W_{11}}}{W_{\max}}\]
\[\color{green}{w_{12}}= \frac{ \color{green}{l_{12} m_{12} n_{12} o_{12}}}{\max\left(l_{ij} m_{ij} n_{ij} o_{ij} \right)} = \frac{ \color{green}{W_{12}}}{W_{\max}}\]
\[\color{red}{w_{22}}= \frac{ \color{red}{l_{22} m_{22} n_{22} o_{22}}}{\max\left(l_{ij} m_{ij} n_{ij} o_{ij} \right)} = \frac{ \color{red}{W_{22}}}{W_{\max}}\]
14.3 Modelando uma geração de seleção
Dada aptidão reprodutiva relativa de cada genótipo e a expectitiva de cruzamento ao acaso do princípio de Hardy-Weinberg, podemos definir a expectativa das frequências genotípicas após uma geração de seleção natural geração como sendo …
… para o alelo \(1\):
\[
\begin{aligned}
p_{t+1}
&= \frac{p^2 N_t \color{blue}{w_{11}} + pq N_t \color{green}{w_{12}}}{p^2 N_t \color{blue}{w_{11}} + 2pq N_t \color{green}{w_{12}} + q^2 N_t \color{red}{w_{22}}} \\
\\
&= \frac{N_t \left( p^2 \color{blue}{w_{11}} + pq \color{green}{w_{12}} \right)}{N_t \left( p^2 \color{blue}{w_{11}} + 2pq \color{green}{w_{12}} + q^2 \color{red}{w_{22}}\right)} \\
\\
&= \frac{p^2 \color{blue}{w_{11}} + pq \color{green}{w_{12}}}{p^2 \color{blue}{w_{11}} + 2pq \color{green}{w_{12}} + q^2 \color{red}{w_{22}}}
\end{aligned}
\]
… e para o alelo \(2\):
\[ \begin{aligned} q_{t+1} &= \frac{q^2 N_t \color{red}{w_{22}} + pq N_t \color{green}{w_{12}}}{p^2 N_t \color{blue}{w_{11}} + 2pq N_t \color{green}{w_{12}} + q^2 N_t \color{red}{w_{22}}} \\ \\ &= \frac{N_t \left( q^2 \color{red}{w_{22}} + pq \color{green}{w_{12}} \right)}{N_t \left( p^2 \color{blue}{w_{11}} + 2pq \color{green}{w_{12}} + q^2 \color{red}{w_{22}}\right)} \\ \\ &= \frac{q^2 \color{red}{w_{22}} + pq \color{green}{w_{12}}}{p^2 \color{blue}{w_{11}} + 2pq \color{green}{w_{12}} + q^2 \color{red}{w_{22}}} \end{aligned} \]
Assim, cada genótipo irá contribuir com genes para a próxima geração ponderadamente a sua aptidão reprodutiva relativa, e a soma da contribuição ponderada de todos os genótipos é definida como a aptidão reprodutiva média da população (\(\bar{w}\)), de forma que:
\[\bar{w} = p^2 w_{11} + 2pq w_{12} + q^2 w_{22}\]
Perceba que \(\bar{w}\) irá diferir da proporção total da população antes da seleção toda vez que (\(w<1\)) para qualquer um dos genótipos, e a diferença entre esse total inicial e a aptidão reprodutiva média (\(1 - \bar{w}\)) irá refletir a parcela da população que foi eliminada pela seleção.
Desta forma, para cada genótipo, temos:
| Parâmetro | \(D(11)\) | \(H(12)\) | \(R(22)\) | Total |
|---|---|---|---|---|
| Aptidão reprodutiva relativa | \(w_{11}\) | \(w_{12}\) | \(w_{22}\) | \(-\) |
| Frequência (\(t\)) | \(p^2\) | \(2pq\) | \(q^2\) | \(1\) |
| Contribuição ponderada | \(p^2 \cdot w_{11}\) | \(2pq \cdot w_{12}\) | \(q^2 \cdot w_{22}\) | \(\bar{w}\) |
| Frequência (\(t+1\)) | \(\frac{p^2 \cdot w_{11}}{\bar{w}}\) | \(\frac{2pq \cdot w_{12}}{\bar{w}}\) | \(\frac{q^2 \cdot w_{22}}{\bar{w}}\) | \(1\) |
Ao padronizar as contribuições genotípicas dos indivíduos, pode-se estimar as freqüências alélicas para a próxima geração como:
\[p_{t+1} = \frac{p^2 w_{11} + pq w_{12}}{\bar{w}}\]
\[q_{t+1} = \frac{q^2 w_{22} + pq w_{12}}{\bar{w}}\]
Assim, a cada geração de seleção natural, é necessário estimar a contribuição ponderada de cada genótipo para a geração seguinte, que dependerá das frequências alélicas que seguem evoluindo ao longo das gerações. E como a aptidão reprodutiva média da população, que relativiza as frequências alélicas após uma geração de seleção, também irá mudar de geração para geração, um modelo geral para estimar as frequências alélicas em uma dada geração \(t\) não é possível de ser estabelecido.
Contudo essa modelagem da seleção natural de uma geração para a outra pode ser repetido passo-a-passo por várias gerações, algoritmo esse que é facilmente calculado em uma planilha eletrônica (ver aqui).
Modelos de seleção natural são melhor compreendidos usando uma medida conhecida como coeficiente de seleção (\(s\)), que é o oposto de aptidão (\(w\)).
Como a aptidão reprodutiva representa o sucesso reprodutivo de um genótipo, enquanto que o coeficiente de seleção corresponde a parcela dos indivíduos que não sobreviveram ou se reproduziram.
O valores de \(w\) e \(s\) podem também ser interpretados como:
- a probabilidade de um indivíduo de um dado genótipo de sobreviver e se reproduzir, ou de ser eliminado por seleção; ou
- a proporção dos alelos que serão passados para a próxima geração, ou que serão removidos do pool gênico por seleção.
As duas medidas estão relacionadas, de forma que:
\[w+s=1\]
Assim, a aptidão reprodutiva pode ser expressa em função do coeficiente de seleção, onde:
\[w=1-s\]
14.4 Tipos de de seleção gênica
Figura 14.4: Hemácias com traços falcêmicos, promovidos pelo alelo HbS da cadeia da beta-globina, apresentam maior resistência à infecções por malária. Este é um polimorfismo balanceado, no qual a seleção natural favorece uma frequência de alelo intermediária devido a um equilíbrio estável de contribuições de aptidão positivas (resistência a malária) e negativas (anemia) para os excessos médios dos alelos polimórficos.
Grosse et al 2011. doi: 10.1016/j.amepre.2011.09.013
Elguero et al 2015. doi: 10.1073/pnas.1505665112
Kato et al 2018. doi: 10.1038/nrdp.2018.10
A depender da aptidão relativa de cada genótipo, quatro tipos de seleção gênica poderão ocorrer:
Perceba que, independentemente dos genótipos favorecidos ou desvantajosos, a seleção natural irá promover o aumento da aptidão reprodutiva média da população, e com o passar das gerações, \(\bar{w} \to 1\).
14.4.1 Seleção contra o recessivo
Também chamada de seleção negativa ou seleção purificadora, esse tipo de seleção elimina polimorfismos deletérios (tipicamente recessivos).
Neste tipo de seleção, a aptidão dos genótipos é expressa como:
\[ \color{green}{w_{11} = w_{12} = 1}\]
\[ \color{red}{w_{22} = 1-s}\]
Em uma grande parte dos casos, envolve alelos letais ou estéreis (\(s=1\)).
Casos de forte seleção negativa produzem uma diminuição no nível de variação em torno do locus sob seleção. A eliminação incidental de alelos não deletérios devido a ligação genética com alelos deletérios é chamada de seleção de fundo (do inglês, backgound selection; Charlesworth et al 1993; Charlesworth 2012, 2013).
Na seleção contra o recessivo:
a frequência do alelo recessivo (\(q\)) diminui rapidamente nas primeiras gerações de seleção, e com o tempo, \(q \to 0\).
a quantidade de mudança evolutiva (\(\Delta q\)) é negativa ao longo das gerações, porque a frequência alélica \(q\) está diminuindo, e com o tempo, (\(\Delta q \to 0\)); e
a aptidão média \(\bar{w}\) da população aumenta a cada geração, e com o tempo, \(\bar{w} \to 1\).
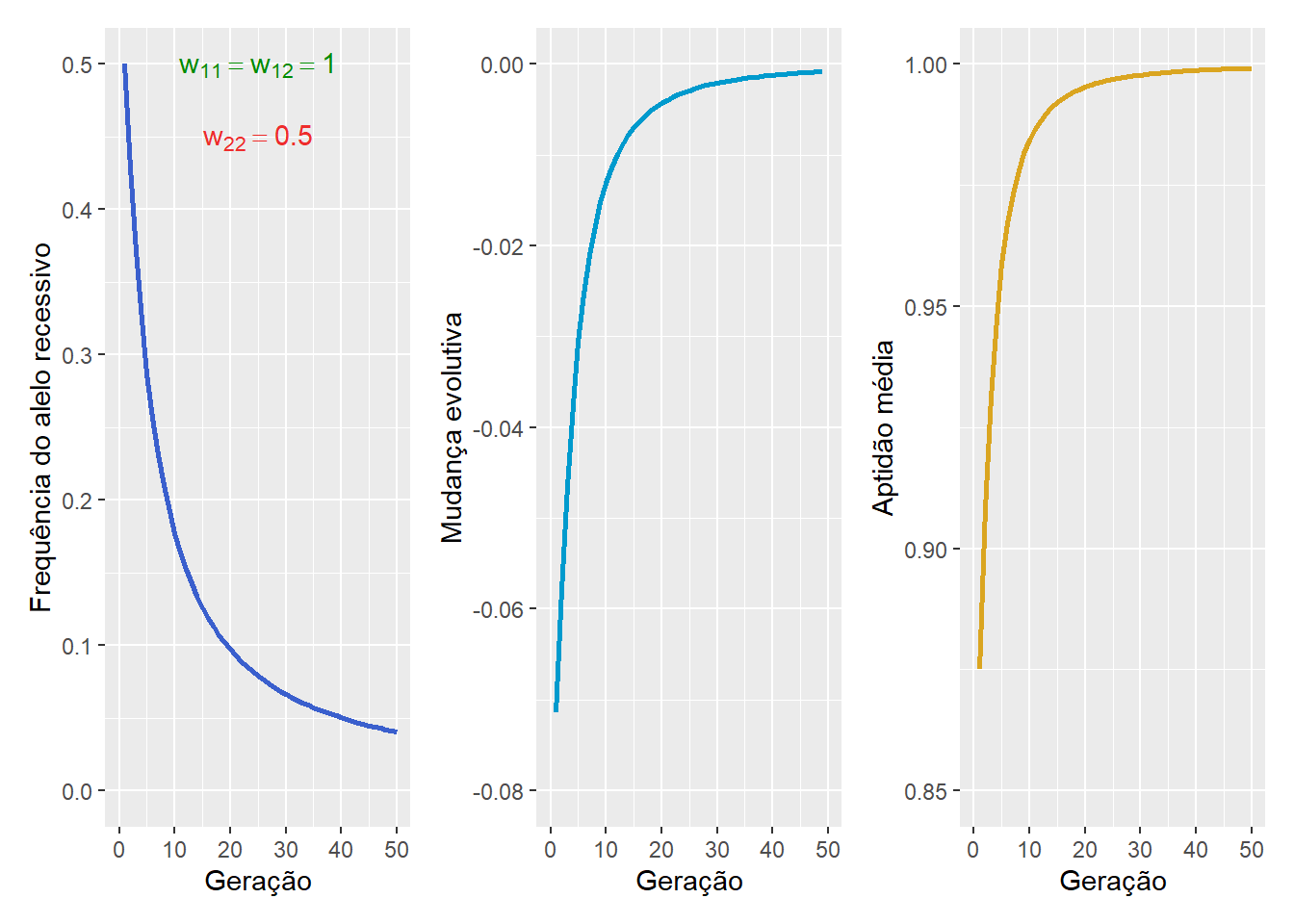
Figura 14.5: A frequência do alelo recessivo (esquerda), a quantidade de mudança evolutiva (centro) e a aptidão média (direita) na seleção contra o recessivo.
14.4.2 Seleção a favor do dominante
Também chamada de seleção positiva ou seleção direcional, esse tipo de seleção favorece o aumento de um determinado alelo na população (tipicamente dominante ou co-dominante).
Neste tipo de seleção, a aptidão relativa dos genótipos pode ser expressa como:
\[ \color{green}{w_{11} = 1}\]
\[ \color{goldenrod}{w_{12} = 1-hs}\]
\[ \color{red}{w_{22} = 1-s}\]
onde \(h\) é o grau de dominância entre os alelos, de forma que o alelo \(1\) será dominante quando \(h=0\), o alelo \(2\) será dominante quando \(h=1\), e os alelos serão codominantes para todo \(0<h<1\).
Perceba que em um locus gênico bialélico com dominãncia completa, a seleção contra o recessivo e a seleção a favor do dominante levarão praticamente ao mesmo tipo de seleção; contudo, em um locus multialélico, a seleção a favor do dominante irá favorecer um determinado alelo em detrimento de outros alelos dominantes e/ou codominantes, fazendo que a a frequência alélica mude em direção ao aumento daquele alelo mais vantajoso.
O aumento da frequência de alelos vantajosos arrasta com ele polimorfismos neutros que estão em regiões gênicas ligadas, promovendo um tipo especial de seleção de fundo conhecido como carona genética (do inglês, genetic hitchhiking; Maynard-Smith & Haigh 1974, Gillespie 2000).
Casos de forte ou rápida seleção positiva são conhecidos como varredura seletiva (do inglês, selective sweep; ver também Hermisson & Pennings 2005 e Pennings & Hermisson 2006).
Assim, na seleção a favor do dominante:
a frequência do alelo dominante (\(p\)) aumenta rapidamente nas primeiras gerações de seleção, e com o tempo, \(p \to 1\).
a quantidade de mudança evolutiva (\(\Delta p\)) é positiva ao longo das gerações, porque a frequência alélica \(p\) está aumentando, mas com o tempo, (\(\Delta p \to 0\)); e
a aptidão relativa média \(\bar{w}\) da população aumenta a cada geração, de forma que \(\bar{w} \to 1\).
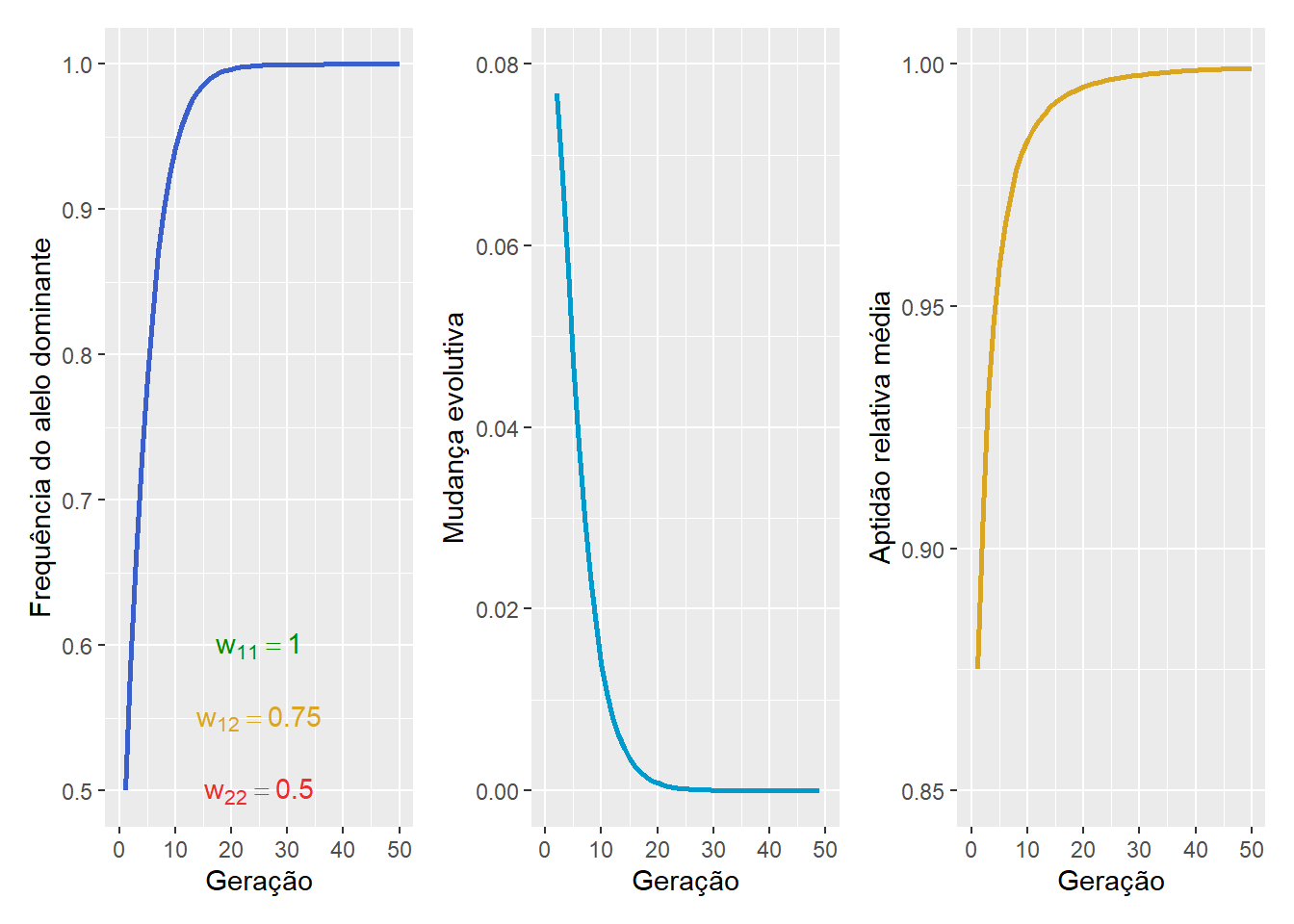
Figura 14.6: A frequência do alelo dominate (esquerda), a quantidade de mudança evolutiva (centro) e a aptidão relativa média (direita) na seleção a favor do dominante.
14.4.3 Seleção a favor do heterozigoto
Também chamada de sobredominância, ocorre quando heterozigotos têm uma aptidão maior do que os homozigotos, de forma que a aptidão relativa dos genótipos sejam:
\[ \color{red}{w_{11} = 1-s_1}\]
\[ \color{green}{w_{12} = 1}\]
\[ \color{red}{w_{22} = 1-s_2}\]
Este processo promove a conservação de polimorfismos genéticos, impedindo que alelos sejam perdidos por deriva genética, sendo assim um processo de seleção balanceadora (do inglês, balancing selection) que leva a um equilíbrio estável das frequências alélicas.
A vantagem do heterozigoto é o principal mecanismo subjacente da heterose (do inglês, heterosis), ou “vigor híbrido”, que é a função melhorada ou aumentada de qualquer qualidade biológica em uma prole híbrida, geralmente por conta do o mascaramento de alelos recessivos deletérios em linhagens puras ou isoladas.
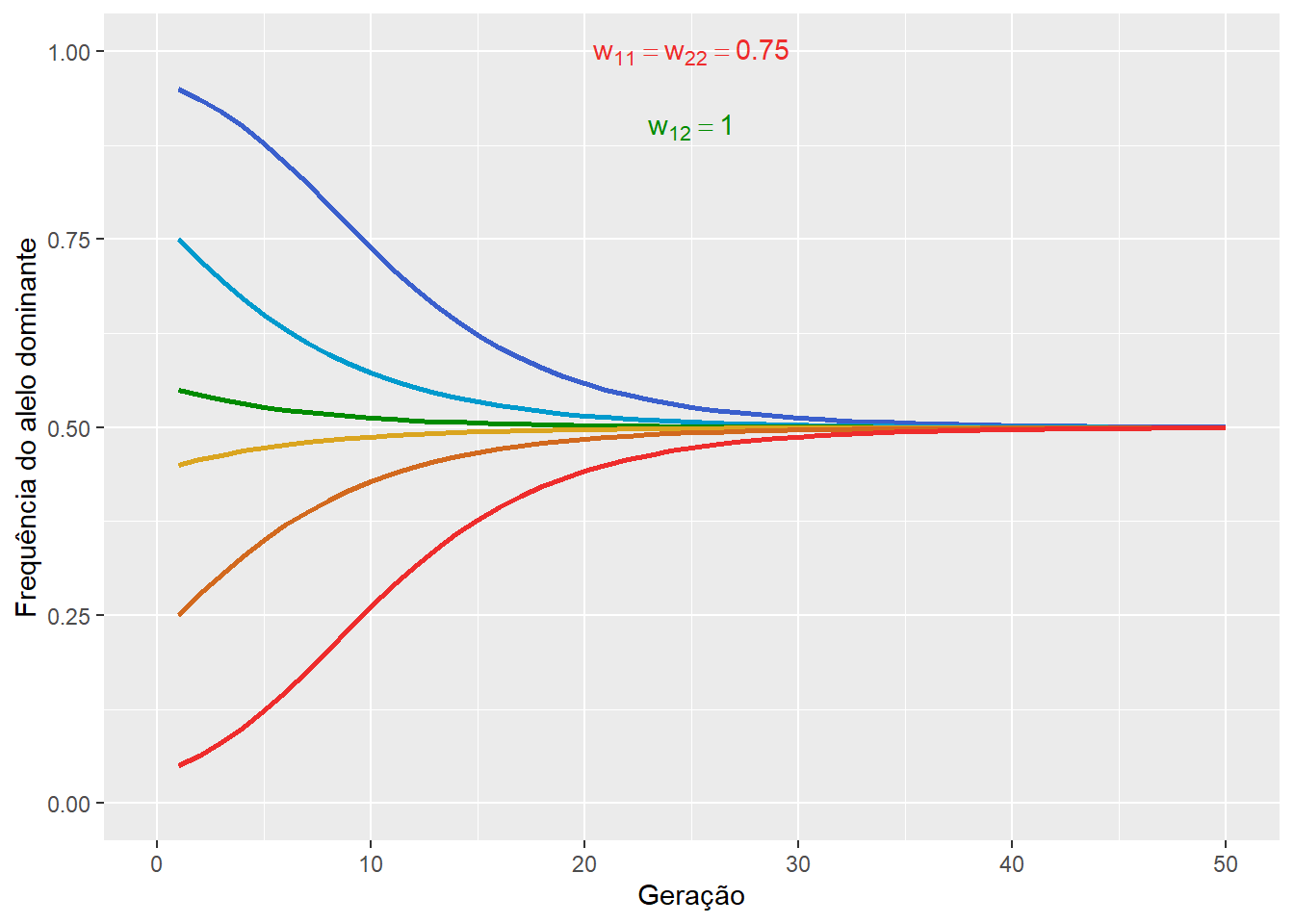
Figura 14.7: A frequência do alelo dominante na seleção a favor do heterozigoto, mostrando o processo de seleção balanceadora estável.
14.4.4 Seleção contra o heterozigoto
Também chamada de subdominância, ocorre quando heterozigotos têm uma aptidão menor do que os homozigotos, de forma que a aptidão dos genótipos sejam, por exemplo:
\[ \color{green}{w_{11} = 1}\]
\[ \color{red}{w_{12} = 1-s}\]
\[ \color{green}{w_{22} = 1}\]
Esse tipo de seleção também promove seleção balanceadora, mas que resulta em um equilíbrio instável das frequências alélicas.
Envolve muitas vezes polimorfismos adaptados a condições locais de diferentes subpopulações, e desta froma, hidridos, ao apresentar fenótipos intermediários, irão ter menor aptidão nos ambientes dos parentais.
O acúmulo dessas incompatibilidades ao longo do tempo pode levar à especiação, como previsto pelo modelo Bateson–Dobzhansky–Muller de isolamento pós-zigótico (Orr 1996).
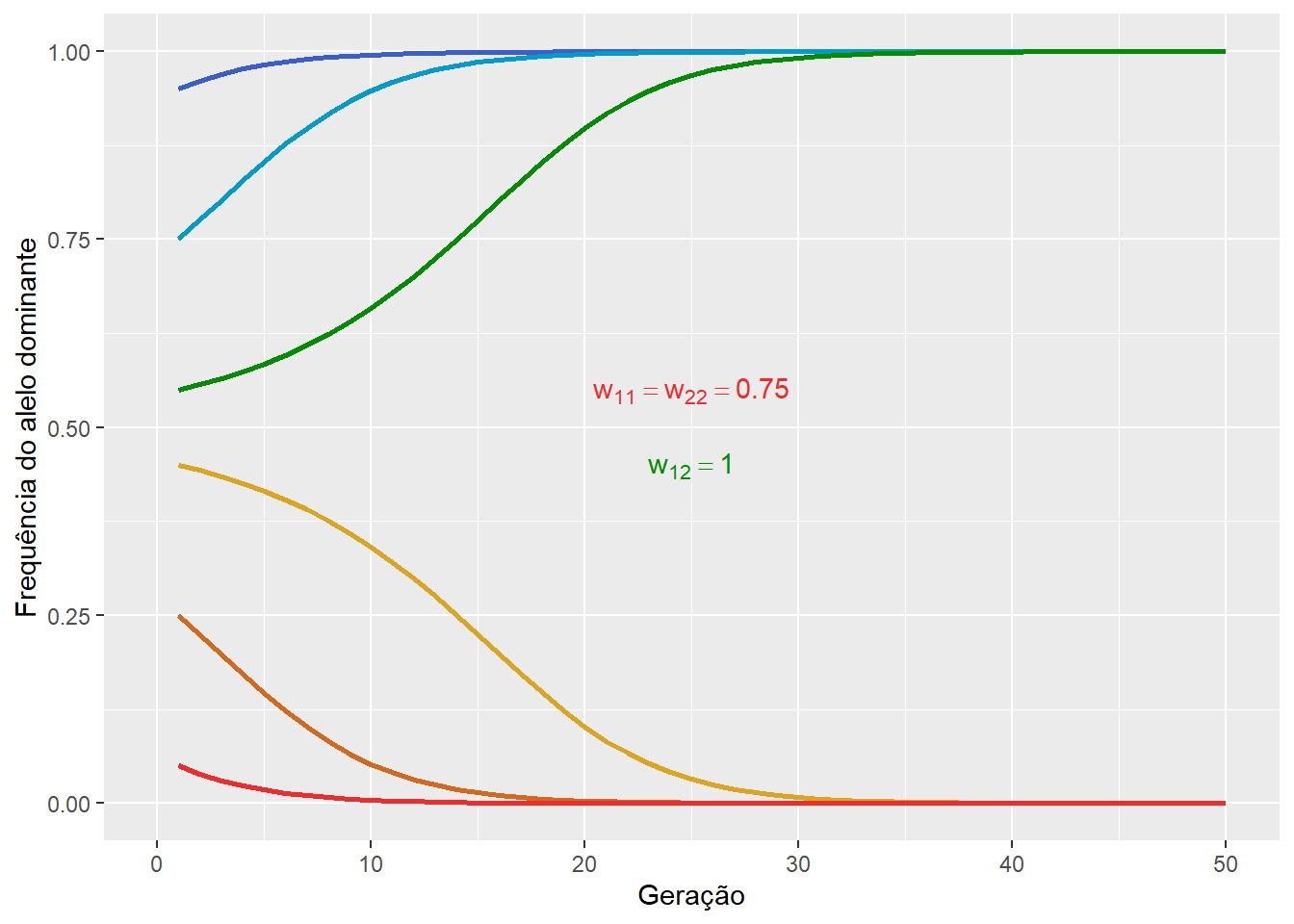
Figura 14.8: A frequência do alelo dominante na seleção contra o heterozigoto, mostrando o processo de seleção balanceadora instável.
Bateson 1909. doi: 10.1017/CBO9780511693953.007
Dobzhansky 1934. doi: 10.1007/BF00374056
Muller 1942. Biology Symposium 6, 71–125
14.5 Seleção em organismos haplóides
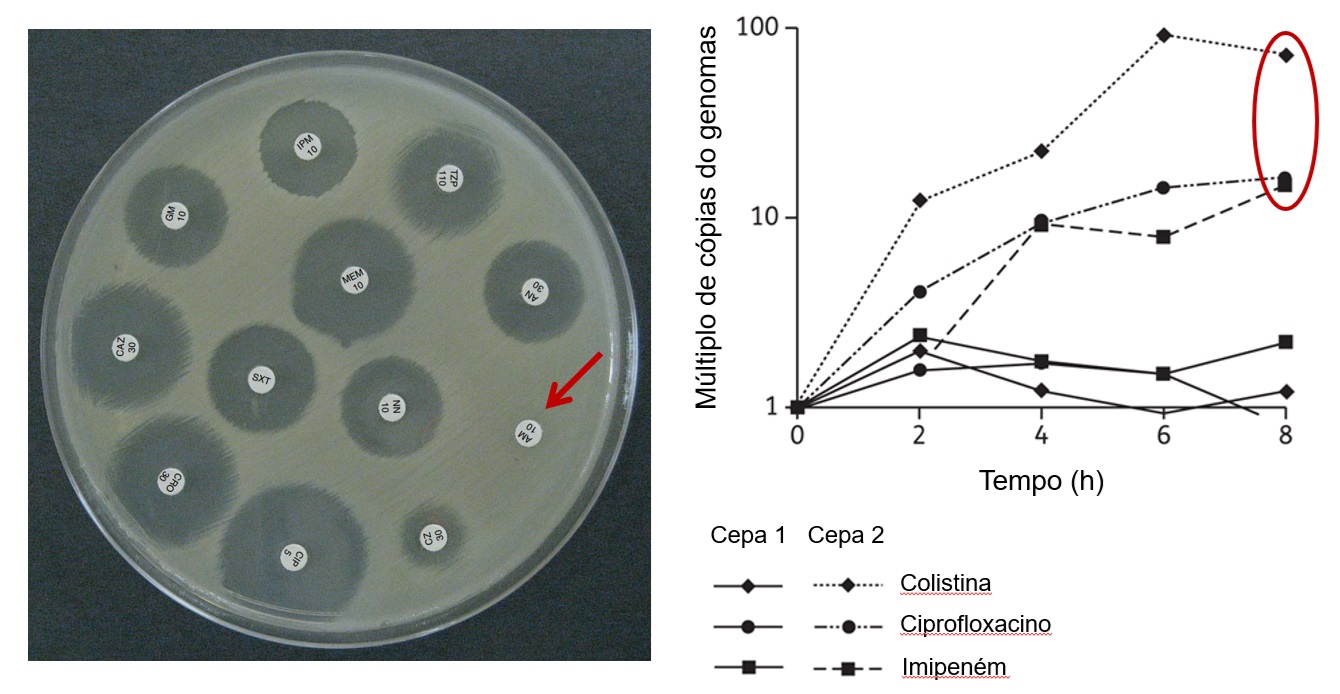
Figura 14.9: A antibioticoterapia apropriada requer a identificação do agente causador da infecção e seu perfil de resistência. No teste de disco difusão para Staphylococcus aureus, a ausência de uma zona de inibição de crescimento em torno do disco de ampicilina (AM) indica resistência ao antibiótico. No PCR em tempo real, a cepa resistente de Acinetobacter baumannii apresenta maior número de cópias de seus genomas.
Tenover 2009. doi: 10.1016/b978-012373944-5.00239-x
Pulido et al. 2013. doi: 10.1093/jac/dkt253
Considere um microorganismo de reprodução assexuada, sem sobreposição de gerações, com um locus gênico com dois genótipos \(\color{#2c89a0}{A}\) e \(\color{#2ca05a}{B}\). Uma vez que são organismos haplóides, as frequências genotípicas e alélicas são iguais, de forma que:
\[f(\color{#2c89a0}{A}) = p \]
\[f(\color{#2ca05a}{B}) = q \]
Se na geração inicial (\(t=0\)) a frequência dos dois genótipos são iguais (\(p = q = 0.5\)), eventos reprodutivos irão promover o nascimento (\(N\)) de novos alelos na população, enquanto que a falha em sobreviver ao ambiente irá levar a morte (\(M\)) desses alelos.
Figura 14.10: Alelos surgem com o nascimento (reprodução) e desaparecem com a morte (sobrevivência) dos organismos haplóides.
Assim,
| \[\color{#2c89a0}{N_A} = 10\] | \[\color{#2ca05a}{N_B} = 10\] |
| \[\color{#2c89a0}{M_A} = 1\] | \[\color{#2ca05a}{M_B} = 4\] |
Desa forma, temos que a aptidão absoluta \(W\) dos genótipos \(\color{#2c89a0}{A}\) e \(\color{#2ca05a}{B}\) será a proporção de sobreviventes, ou não-mortos (\(N-M\)), e nascimentos (\(N\)), sendo:
| \[ \begin{aligned} \color{#2c89a0}{W_A} &= \frac{\color{#2c89a0}{N_A}-\color{#2c89a0}{M_A}}{\color{#2c89a0}{N_A}} \\ &= \frac{10-1}{10} \\ &= \frac{9}{10} \\ &= 0.9 \end{aligned} \] | \[ \begin{aligned} \color{#2ca05a}{W_B} &= \frac{\color{#2ca05a}{N_B}-\color{#2ca05a}{M_B}}{\color{#2ca05a}{N_B}} \\ &= \frac{10-4}{10} \\ &= \frac{6}{10} \\ &= 0.6 \end{aligned} \] |
E assim podemos obter a aptidão relativa \(w\)…
| \[ \begin{aligned} \color{#2c89a0}{w_A} &= \frac{\color{#2c89a0}{W_A}}{W_{max}} \\ &= \frac{\color{#2c89a0}{W_A}}{\color{#2c89a0}{W_A}} \\ &= \frac{0.9}{0.9} \\ &= 1 \end{aligned} \] | \[ \begin{aligned} \color{#2ca05a}{w_B} &= \frac{\color{#2ca05a}{W_B}}{W_{max}} \\ &= \frac{\color{#2ca05a}{W_B}}{\color{#2c89a0}{W_A}} \\ &= \frac{0.6}{0.9} \\ &\approx 0.667 \end{aligned} \] |
… e o coeficiente de seleção \(s\) dos genótipos.
| \[ \begin{aligned} \color{#2c89a0}{s_A} &= 1-\color{#2c89a0}{w_A} \\ &= 1-1 \\ &= 0 \end{aligned} \] | \[ \begin{aligned} \color{#2ca05a}{s_B} &= 1-\color{#2ca05a}{w_B} \\ &\approx 1 - 0.667 \\ &\approx 0.333 \end{aligned} \] |
Desta forma, dos 20 alelos nascidos na população, somente 15 sobreviveram para serem transmitidos para a próxima geração. E assim, a frequência dos genótipos no próxima geração \(t=1\) serão:
| \[ \begin{aligned} \color{#2c89a0}{p_{1}} &= \frac{9}{15} \\ &= 0.6 \end{aligned} \] | \[ \begin{aligned} \color{#2ca05a}{q_{1}} &= \frac{6}{15} \\ &= 0.4 \end{aligned} \] |
As frequências genotípicas podem ser expressas em termos das frequências iniciais dos alelos \(\color{#2c89a0}{p_0}\) e \(\color{#2ca05a}{q_0}\) e dos valores de aptidão relativa de cada genótipo, \(\color{#2c89a0}{w_M}\) e \(\color{#2ca05a}{w_N}\), de forma que:
| \[\color{#2c89a0}{p_{1}} = \frac{\color{#2c89a0}{p_{0}}\color{#2c89a0}{w_A}}{\color{#2c89a0}{p_{0}}\color{#2c89a0}{w_A}+\color{#2ca05a}{q_{0}}\color{#2ca05a}{w_B}}\] | \[\color{#2ca05a}{q_{1}} = \frac{\color{#2ca05a}{q_{0}}\color{#2ca05a}{w_B}}{\color{#2c89a0}{p_{0}}\color{#2c89a0}{w_A}+\color{#2ca05a}{q_{0}}\color{#2ca05a}{w_B}}\] |
Uma vez que \(w = 1-s\), podemos substituir os termos nas equações acima de forma que:
| \[ \color{#2c89a0}{p_{1}} = \frac{\color{#2c89a0}{p_{0}}(1-\color{#2c89a0}{s_A})}{\color{#2c89a0}{p_{0}}(1-\color{#2c89a0}{s_A})+\color{#2ca05a}{q_{0}}(1-\color{#2ca05a}{s_B})} \] | \[ \color{#2ca05a}{q_{1}} = \frac{\color{#2ca05a}{q_{0}}(1-\color{#2ca05a}{s_B})}{\color{#2c89a0}{p_{0}}(1-\color{#2c89a0}{s_A})+\color{#2ca05a}{q_{0}}(1-\color{#2ca05a}{s_B})} \] |
E sabendo que \(\color{#2c89a0}{s_A}=0\), teremos:
| \[ \begin{aligned} \color{#2c89a0}{p_{1}} &= \frac{\color{#2c89a0}{p_{0}}}{\color{#2c89a0}{p_{0}}+\color{#2ca05a}{q_{0}}-\color{#2ca05a}{q_{0}}\color{#2ca05a}{s_B}} \\ &= \frac{\color{#2c89a0}{p_{0}}}{1-\color{#2ca05a}{q_{0}}\color{#2ca05a}{s_B}} \\ \end{aligned} \] | \[ \begin{aligned} \color{#2ca05a}{q_{1}} &= \frac{\color{#2ca05a}{q_{0}}(1-\color{#2ca05a}{s_B})}{\color{#2c89a0}{p_{0}}+\color{#2ca05a}{q_{0}}-\color{#2ca05a}{q_{0}}\color{#2ca05a}{s_B}} \\ &= \frac{\color{#2ca05a}{q_{0}}(1-\color{#2ca05a}{s_B})}{1-\color{#2ca05a}{q_{0}}\color{#2ca05a}{s_B}} \\ \end{aligned} \] |
Considerando que não haja variação no ambiente, de forma que os valores de coeficiente de seleção (\(s\)) permaneçam estáveis ao longo das gerações, teremos que as frequências genotípicas na geração seguinte (\(t=2\)) será:
| \[ \begin{aligned} \color{#2c89a0}{p_{2}} &= \frac{\color{#2c89a0}{p_{1}}}{1-\color{#2ca05a}{q_{1}}\color{#2ca05a}{s_B}} \\ &= \frac{0.6}{1-(0.4 \times 0.333)} \\ &\approx 0.692 \end{aligned} \] | \[ \begin{aligned} \color{#2ca05a}{q_{2}} &= \frac{\color{#2ca05a}{q_{1}}(1-\color{#2ca05a}{s_B})}{1-\color{#2ca05a}{q_{1}}\color{#2ca05a}{s_B}} \\ &= \frac{0.4(1-0.333)}{1-0.4 \times 0.333} \\ &\approx 0.308 \end{aligned} \] |
E na geração subsequente (\(t=3\)) teremos:
| \[ \begin{aligned} \color{#2c89a0}{p_{3}} &= \frac{\color{#2c89a0}{p_{2}}}{1-\color{#2ca05a}{q_{2}}\color{#2ca05a}{s_B}} \\ &\approx \frac{0.692}{1-(0.308 \times 0.333)} \\ &\approx 0.771 \end{aligned} \] | \[ \begin{aligned} \color{#2ca05a}{q_{3}} &= \frac{\color{#2ca05a}{q_{2}}(1-\color{#2ca05a}{s_B})}{1-\color{#2ca05a}{q_{2}}\color{#2ca05a}{s_B}} \\ &\approx \frac{0.308(1-0.333)}{1-0.308 \times 0.333} \\ &\approx 0.229 \end{aligned} \] |
Figura 14.11: A frequência dos alelos evolui por seleção nos organismos haplóides proporcionalmente ao coeficiente de seleção (\(s\)) do genótipo \(N\), menos adaptado, que tende a ser substituído na população pelo genótipo \(M\), mais adaptado.
Se o crescimento populacional bacteriano segue um modelo geométrico, de forma que:
\[ N_t = N_0 \left( 1 + r\right)^t \] onde
\(N_t\) é o tamanho da população na geração \(t\);
\(N_0\) é o tamanho inicial da população (\(t=0\));
\(r\) é a taxa de crescimento instrinseca da população; e
\(t\) é o tempo transcorrido, em número de gerações.
Podemos considerar o crescimento dos genótipos \(\color{#2c89a0}{A}\) e \(\color{#2ca05a}{B}\) de modo que:
| \[ \color{#2c89a0}{A_t} = \color{#2c89a0}{A_0} \left( 1 + \color{#2c89a0}{a}\right)^t \] | \[ \color{#2ca05a}{B_t} = \color{#2ca05a}{B_0} \left( 1 + \color{#2ca05a}{b}\right)^t \] |
onde, respectivamente aos genótipos \(\color{#2c89a0}{A}\) e \(\color{#2ca05a}{B}\),
\(\color{#2c89a0}{A_t}\) e \(\color{#2ca05a}{B_t}\) são o número de indivíduos na geração \(t\);
\(\color{#2c89a0}{A_0}\) e \(\color{#2ca05a}{B_0}\) são o número de indivíduos iniciais (\(t=0\)) de cada genótipo;
\(\color{#2c89a0}{a}\) e \(\color{#2ca05a}{b}\) são as taxas de crescimento instrinsecas de cada genótipo; e
\(t\) continua sendo o número de gerações.
Assim:
Se \(\color{#2c89a0}{a}=\color{#2ca05a}{b}\), ambos genótipos crescem na mesma proporção, e não há seleção.
Se \(\color{#2c89a0}{a} \ne \color{#2ca05a}{b}\), um genótipo cresce proporcionalmente mais rápido do que outro, e há seleção.
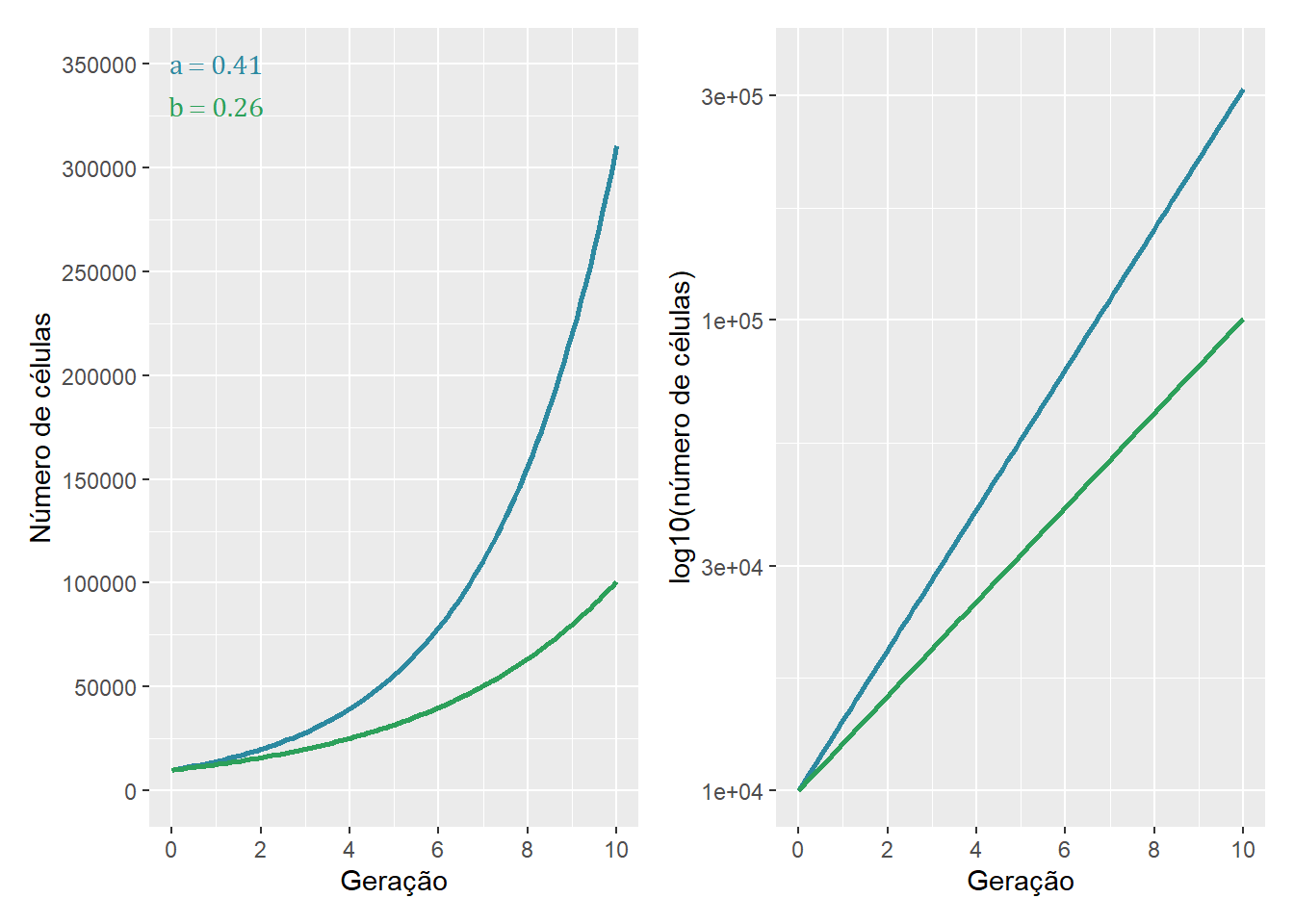
Figura 14.12: As populações de células de duas cepas bacterianas M e N, com taxas de crescimentos instrínsecas m e n aumentam de tamanho exponencialmente, mas a cepa M aumenta mais rapidamente do que a cepa N por seleção.
Assim, o coeficiente de seleção (\(s\)) entre as cepas deve ser dado pela proporção com que as cepas \(\color{#2c89a0}{A}\) e \(\color{#2ca05a}{B}\) crescem em relação uma com a outra.
A razão do número de células da cepa \(\color{#2c89a0}{A}\) para a \(\color{#2ca05a}{B}\) no tempo \(t\) é dada por:
\[ \frac{\color{#2c89a0}{A_t}}{\color{#2ca05a}{B_t}} = \frac{\color{#2c89a0}{A_0}\left(1+\color{#2c89a0}{a} \right)^t}{\color{#2ca05a}{B_0}\left(1+\color{#2ca05a}{b} \right)^t} \] Ou
\[ \frac{\color{#2c89a0}{A_t}}{\color{#2ca05a}{B_t}} = \frac{\color{#2c89a0}{A_0}}{\color{#2ca05a}{B_0}} \times \left( \frac{1+\color{#2c89a0}{a}}{1+\color{#2ca05a}{b}} \right)^t \]
Se ao invés do número de células das cepas \(\color{#2c89a0}{A}\) e \(\color{#2ca05a}{B}\) nos interessarmos pelas frequências alélicas \(\color{#2c89a0}{p}\) e \(\color{#2ca05a}{q}\) respectivamente, teríamos que:
\[ \frac{\color{#2c89a0}{p_t}}{\color{#2ca05a}{q_t}} = \frac{\color{#2c89a0}{p_0}}{\color{#2ca05a}{q_0}} \times \left( \frac{1+\color{#2c89a0}{a}}{1+\color{#2ca05a}{b}} \right)^t \]
Sendo \(w\) a aptidão relativa entre as cepas \(\color{#2c89a0}{A}\) e \(\color{#2ca05a}{B}\), onde:
\[ w = \left( \frac{1+\color{#2c89a0}{a}}{1+\color{#2ca05a}{b}} \right) \]
Temos que:
\[ \frac{\color{#2c89a0}{p_t}}{\color{#2ca05a}{q_t}} = \frac{\color{#2c89a0}{p_0}}{\color{#2ca05a}{q_0}} \times w^t \] De forma que, a relação entre as frequências dos dois genótipos após \(t\) gerações vai depender de suas frequências iniciais e da razão entre suas taxas de crescimento instrínsecas, i.e., da aptidão relativa entre os genótipos \(\color{#2c89a0}{A}\) e \(\color{#2ca05a}{B}\).
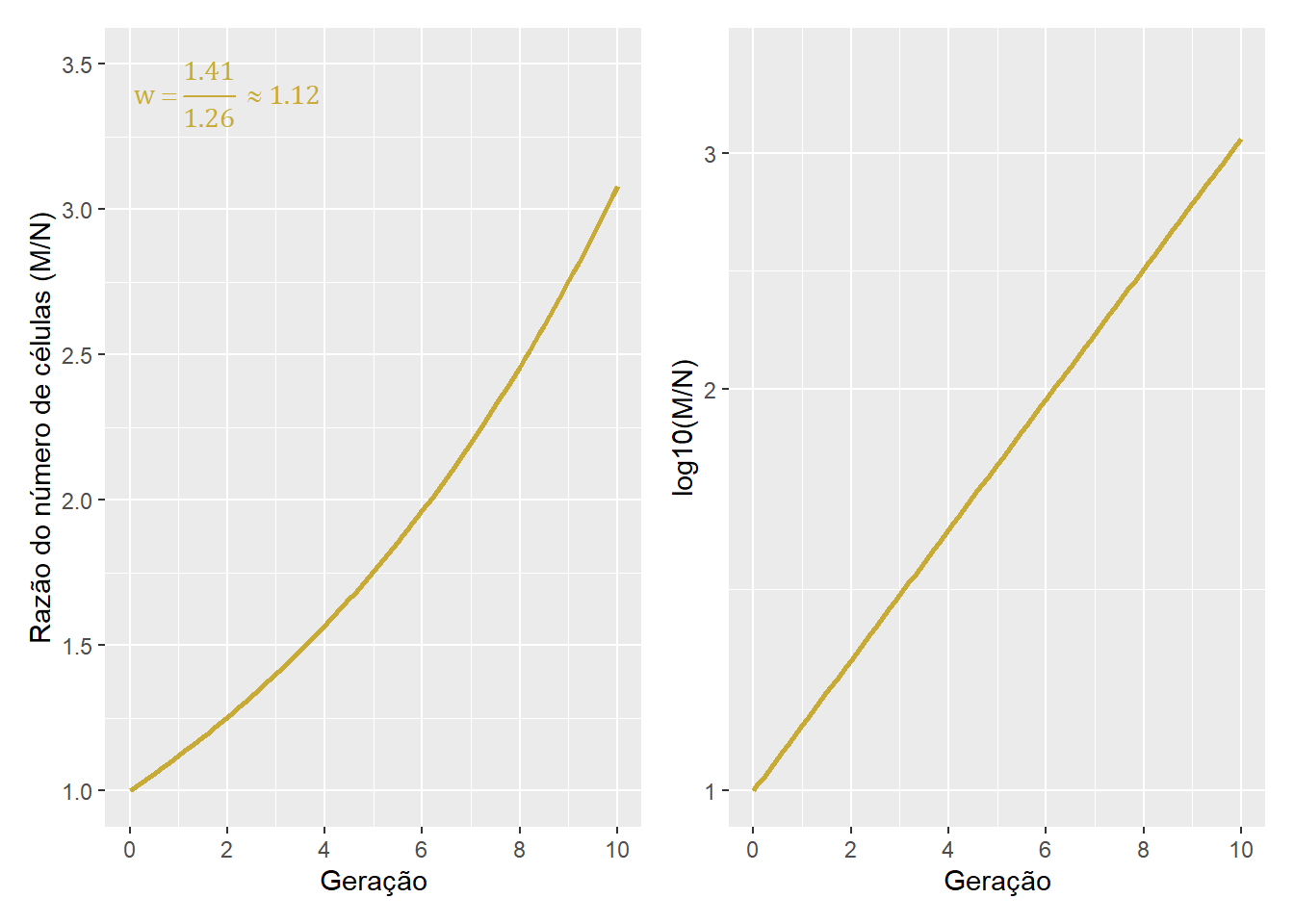
Figura 14.13: A cepa \(M\) se reproduz \(w\) vezes mais rápido do que a cepa \(N\).
14.6 Paisagem Adaptativa
).](figs/wright1932.jpg)
Figura 14.14: A evolução da aptidão de uma população pode ser estudada visualizando a distribuição dos valores de aptidão de diferentes genótipos como um tipo de paisagem. Retirado de Wright (1932).
Wright S. (1932) The roles of mutation, inbreeding, crossbreeding and selection in evolution. Proceedings of the Sixth International Congress on Genetics 1, 356–366.
Se presumirmos que todo genótipo tenha uma aptidão bem definida, genótipos semelhantes serão considerados “próximos” uns dos outros e apresentarão aptidões semelhantes, enquanto que genótipos muito diferentes estarão “distantes” uns dos outros e tenderão a diferir em aptidão.
Assim, a paisagem adaptativa será o conjunto de todos os genótipos possíveis, seu grau de similaridade e seus valores de aptidão relacionados.
As paisagens adaptativas são frequentemente concebidas como cadeias de montanhas, onde:
Picos adaptativos são pontos a partir dos quais todos os caminhos levam para baixo, i.e., diminuem a aptidão.
Vales adptativos são pontos a partir dos quais todos caminhos levam para cima, i.e., aumentam a aptidão.
Trajetória evolutiva é a série de mudanças evolutivas em uma população, normalmente, “morro” acima na paisagem adaptativa. Dado tempo suficiente, um pico evolutivo deverá ser alcançado.
Figura 14.15: Uma vez que a trajetória evolutiva ocorre gradualmente, ao atingir picos (p) adaptativos locais (A e C), as populações deixam de evoluir, mesmo havendo um pico adaptativo global (B) para determinado genótipo, uma vez que ela não consegue atravessar os vales (v) adaptativos.
Diferentes trajetórias evolutivas podem atingir diferentes picos adaptativos, mesmo a partir do mesmo genótipo inicial.
Paisagens adaptativas suaves, com um único pico e sem interações adaptativas entre genótipos, fazem com que diferentes trajetórias evolutivas levem a um mesmo pico adaptativo.
Por outro lado, paisagens adaptativas acidentadas, com vários picos e interações entre genótipos, podem fazer com que diferentes trajetórias evolutivas atinjam diferentes picos adaptativos, mesmo a partir do mesmo genótipo inicial.
).](figs/landscape_roughness.jpg)
Figura 14.16: Diferentes trajetórias evolutivas podem atingir diferentes picos adaptativos, mesmo a partir do mesmo genótipo inicial. Adaptado de Van Cleve & Weissman (2015).
Em um cenário onde o ambiente é estável, a aptidão é igualmente estática, e a população evolui para apenas um pico. Contudo a evolução em ambientes alternados que modulam a paisagem adaptativa pode levar ao cruzamento de vales de aptidão. Á medida que a paisagem adaptativa muda, a população evolui para acompanhar os picos.
).](figs/landscape_evolution.jpg)
Figura 14.17: As mudanças ambientais fazem a ponte entre os vales evolutivos. A. Uma população (círculo) em um ótimo local, separado do ótimo global por um vale adaptativo. B. Uma mudança no ambiente altera a paisagem de forma que a evolução traz a população um novo ótimo, que anteriormente residia no vale. C.O retorno do ambiente para a condição original faz com que a população resida no vale, e que agora pode evoluir para o ótimo global. Adaptado de Steinberg & Ostermeier (2016).
14.7 Equilíbrio mutação-seleção
Figura 14.18: A maioria das mutações são neutras (N), deletérias (D) ou letais (L); mutações vantajosas (V) são relativamente raras.
Sanjuan et al. 2004. doi: 10.1073/pnas.0400146101
Eyre-Walker & Keightley 2007. doi: 10.1038/nrg2146
O influxo de mutações deletérias (e letais) em uma população ao longo do tempo é contrabalançado pela seleção negativa, que age para eliminar essas mutações.
Deixando de lado outros fatores, o número de alelos deletérios é então determinado por um equilíbrio entre a taxa de mutação deletéria e a taxa com que a seleção elimina essas mutações.
Em uma população diplóide, um alelo deletério \(B\) pode ter efeitos diferentes sobre a aptidão individual em heterozigotos \(AB\) e homozigotos \(BB\) dependendo do grau de dominância \(h\) do alelo normal \(A\).
Para representar tal efeito, consideraremos a aptidão relativa de homozigotos deletérios (\(BB\)), como sendo:
\[w_{BB} = 1-s\]
E heterozigotos (\(AB\)) como sendo:
\[w_{AB} = 1-hs\]
Onde \(s\) é o coeficiente de seleção contra o genótipo homozigoto \(BB\), e \(h\) é o grau de dominância do alelo \(A\), variando de 0 (dominância completa) a 1 (recessividade completa).
O grau de dominância afeta a importância relativa da seleção de heterozigotos contra homozigotos.
Se \(\mu \rightarrow 0\), i.e., se a taxa de mutação deletéria é muito baixa, os heterozigotos irão conter a grande maioria dos alelos deletérios \(B\) da população.
Se \(h \ne 0\), i.e., se o alelo \(A\) não é completamente dominante, as mutações deletérias serão primariamente removidas pela seleção negativa dos heterozigotos.
Neste caso:
A mutação converte homozigotos \(AA\) em heterozigotos \(AB\) em uma taxa de mutação \(\mu\); e
A seleção remove heterozigotos com coeficiente de seleção \(hs\).
Assim, a frequência do alelo deletério \(B\) no equilíbrio ($) será:
\[ \hat{q} = \frac{\mu}{hs} \]
Se \(h=0\), i.e., o alelo \(A\) apresenta dominância completa, então o alelo deletério \(B\) só será removido pela seleção dos homozigotos \(BB\).
Neste caso, a frequência \(p\) do alelo normal \(A\) aumenta devido à eliminação seletiva de homozigotos recessivos, de forma que:
\[ \Delta p = \frac{1}{1-sp_{BB}} \] Onde,
\(s\) é o coeficiente de seleção contra o genótipo homozigoto \(BB\); e
\(p_{BB}\) é a frequência denotípica do homozigoto \(BB\).
Por outro lado, a mutação faz com que a frequência \(p\) do alelo normal \(A\) diminua proporcionalmente, e se ignorarmos as mutações reversas, uma vez que estas são muito raras, teremos que:
\[
\Delta p = 1-\mu
\]
Sendo assim, a frequência do genótipo recessivo \(BB\) no equilíbrio \(\hat{p}_{BB}\) entre seleção e mutação será:
\[ \hat{p}_{BB} \approx \frac{\mu}{s} \]
E a freqüência \(\hat{q}\) de alelos deletérios \(B\) será:
\[
\hat{q} \approx \sqrt{\frac{\mu}{s}}
\]
Esta freqüência de equilíbrio é substancialmente maior do que no caso de dominância parcial, porque heterozigotos serão portadores de um grande número de alelos mutantes, e protegidos da seleção.
14.8 Outros tipos de seleção
14.8.1 Conflito intragenômico
A evolução por conflito intragenômico diz respeito a genes com efeitos fenotípicos que promovem sua própria transmissão em detrimento da transmissão de outros genes no mesmo genoma.
Existem vários tipos de elementos genéticos egoístas (SGEs, do inglês selfish genetic elements) que proporcionam conflito genético e impulsionam inovação evolutiva, como transposons e outros elementos transponíveis.
Um outro tipo de conflito genético é o conflito intragenômicos promovido pelo impulso meiótico (meiotic drive), em que um ou mais loci de um genoma manipulam o processo meiótico para favorecer sua transmissão em detrimento de outros. Esse desequilíbrio pode ser desencadeado tanto pela assimetria da meiose ou letalidade de gametas que carregam o alelo/cromossomo em conflito.
Figura 14.19: Exemplo de conflito intragenômico. Um gene ligado ao X pode afetar sua própria transmissão para a próxima geração se puder aumentar a proporção de células de esperma que carregam um cromossomo X em vez de um cromossomo Y, e vice-versa.
Gershenson 1928. doi: 10.1093/genetics/13.6.488
Rhoades 1941. doi: 10.1093/genetics/27.4.395
Sandler & Novitski 1957. doi: 10.1086/281969
Dawkins 1976. ISBN: 9780191093067
Werren 2011. NCBI Books: NBK424883
14.8.2 Seleção de parentesco
A seleção de parentesco favorece o sucesso reprodutivo dos parentes de um organismo, mesmo que custe a própria sobrevivência e reprodução do organismo.
Do ponto de vista de um gene, a aptidão depende, em última análise, somente de deixar para a próxima geração o maior número de cópias de si mesmo na população. Como indivíduos aparentados apresentam (em parte) genes idênticos por descendência entre si, para esses genes, não importa quem está de fato se reproduzindo, contanto que haja suceso reprodutivo.
Assim, os indivíduos que apresentem comportamentos altruístas para com outros aparentados a ele podem aumentar a chance de transmissão de seus genes se beneficiar um número equivalente de descendentes a sua aptidão pessoal.
Aptidão pessoal é o número de descendentes que um indivíduo gera, independentemente de qual indivíduo resgata, cria, ou suporta aquela prole.
Aptidão inclusiva é o número de equivalentes de descendentes que um indivíduo cria, resgata, ou de outra forma, sustenta por meio de seu comportamento, independentemente de quem os gera por reprodução.
Em organismos diplóides, os descendentes diretos de um indivíduo carregam \(1/2\) dos seus genes, definindo o que chamamos de equivalente de descendente. Assim, um sobrinho (filho de seu irmão) carrega \(1/4\) dos seus genes, possuindo \(1/2\) de equivalente de descendente. Da mesma forma, um primo de segundo grau (o filho de seu primo) tem 1/16 dos seus genes, e tem 1/8 de equivalente de descendente. Logo, para os genes, dois sobrinhos, ou oito primos de segundo grau, equivalem a um filho.
De acordo com a regra de Hamilton, os genes associados a comportamentos altruístas aumentarão em frequência quando:
\[ rB > C \]
Onde,
\(r\) é a relação de parentesco do destinatário com o ator,
\(B\) é o benefício reprodutivo obtido pelo destinatário do ato altruísta, e,
\(C\) é o custo reprodutivo para o ator do ato altruísta.
Um exemplo clássico de de seleção de parentesco ocorre nos cães-de-pradaria (Cynomys ludovicianus), os quai mais propensos a dar chamados de alarme se seu grupo doméstico incluir indivíduos aparentados.
Hamilton 1964. doi: 10.1016/0022-5193(64)90038-4
Hamilton 1964. doi: 10.1016/0022-5193(64)90039-6
Birch 2016. doi: 10.1086/687869
14.8.3 Efeito barba-verde
O efeito barba-verde é apresentado em um experimento mental onde a distinção entre reconhecimento/discriminação de parentesco é uma “barba verde” hipotética, onde se imagina que um gene para comportamento social também causa um fenotípico distintivo que pode ser reconhecido por outros portadores do gene.
Assim, formas de auto-reconhecimento genético permitem que o gene em um indivíduo direcione benefícios para outros indivíduos que possuem o mesmo gene.
Por exemplo, o número de rainhas por colônia de formigas-de-fogo (Solenopsis invicta) parece estar associado a genótipos operários no gene de proteína de ligação a feromônio Gp-9.
Figura 14.20: Um portador de um alelo de comportamento direciona o altruísmo para um portador de um alelo de barba que, devido ao desequilíbrio de ligação entre os dois loci, também é provável que seja um portador do alelo de comportamento. Adaptado de Gardner (2019)
Keller & Ross 1998. doi: 10.1038/29064
Krieger & Ross 2002. doi: 10.1126/science.1065247
Krieger & Ross 2005. doi: 10.1093/molbev/msi203
Gardner 2019. doi: 10.1016/j.cub.2019.03.063
14.9 Exercícios
14.9.1 Exercício 1 – Resistência a inseticidas
A resistência a inseticidas organofosfatados e carbamatados em espécies de mosquitos Culex e Anopheles pode ser mediada por quatro mutações independentes no gene da acetilcolinesterase (ace-1). As mutações, resultando em G119S (uma glicina, na posição 119, substituída por uma serina), tornam a enzima insensível à inibição por essses inseticidas (Weill et al 2004).
Desta forma, responda:
Que tipo de seleção essa variante mutante irá promover?
Se os mosquitos que possuem esse alelo de resistência pudessem sobreviver e se reproduzir 10 vezes mais do que os que possuem o alelo sensível, quanto tempo levaria para que a frequência do alelo mudasse de \(p_{G119S} = 0.01\) para \(p_{G119S} = 0.5\)?
14.9.2 Exercício 2 – Equilíbrio mutação-seleção
Imagine um alelo que seja letal em homozigose (deletério) em uma população de Drosophila. Suponha que esse alelo seja mantido por equilíbrio mutação-seleção com \(\mu = 5 \times 10^{-6}\). Calcule a frequência do alelo para:
Um recessivo completo.
Um dominante parcial, quando o grau de dominância é \(h = 0.025\).